Distemper
Diagnóstico de distemper en el laboratorio clínico veterinario
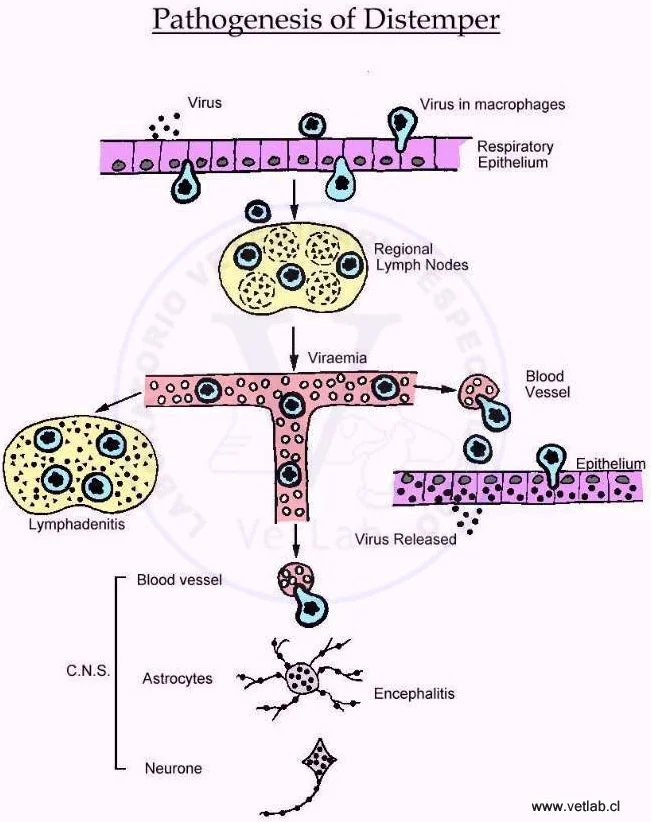
En general, el diagnóstico de la enfermeded por el virus Distemper (CDV), suele limitarse a la sospecha clínica. Si bien la enfermedad multisistémica es fácil de reconocer, no siempre se presenta de esta forma y, para optimizar de las pruebas de laboratorio, se requiere de la comprensión integral de la patogénesis de la enfermedad.
Existe una gran variedad de ensayos clínicos para el diagnóstico ante mortem definitivo del distemper. Sin embargo, debido su curso impredecible (duración variable de la viremia y una respuesta inmune tardía o nula), el diagnóstico definitivo puede ser incierto.
No obstante, la información clínico-patológica puede complementarse con la presencia de anticuerpos. Por ejemplo, la presencia de anticuerpos anti-CDV en el fluído cerebroespinal (FCE), es patognomónica de la encefalitis por Distemper, pues estos anticuerpos se producen en forma local y no están presentes en perros vacunados.
Los exámenes hematológicos y bioquímicos son inespecíficos. Sólo tienen valor para el monitoreo y el pronóstico de la enfermedad.
Suele observarse anemia normocítica -normocrómica y regenerativa, la que puede incrementarse con el curso de la enfermedad.
La linfopenia absoluta ocurre por necrosis del dejido linfoide. Es dependiente de la edad y condición clínica del paciente, así como de la virulencia de la cepa viral.
Trombocitopenia moderada. También es dependiente de la edad, de la condición clínica de paciente y del genotipo viral.
Monocitosis relativa o absoluta es el cambio más constante en el leucograma.
Puede observarse leucocitosis neutrófila con o sin desviación a la izquierda, principalmente en cuadros con sobreinfección bacteriana.
Las proteínas totales generalmente son normales. Pueden aumentar en cuadros que cursan con deshidratación (↑ albúminas)
Las globulinas suelen estar aumentadas en pacientes inmunocompetentes.
En el FCE el diagnóstico puede hacerse si hay aumento de la proteinorraquia, pleocitosis linfocitaria, y son detectados anticuerpos específicos en una muestra no contaminada con sangre periférica.
La sensibilidad de estas pruebas es altamente dependiente de temporización del cuadro clínico.
Las partículas virales (antígenos) se pueden encontrar en los leucocitos, eritrocitos y células epiteliales tempranamente en el curso de la infección, a menudo antes de la aparición de los signos clínicos.
Luego de la aparición de estos signos, el antígeno CDV puede desaparecer de la sangre, conjuntiva y epitelio genital antes de una semana, detectándose por más tiempo en los macrófagos y en el epitelio del tracto respiratorio bajo.
La detección de antígenos virales de Distemper (acúmulos de cápsides intracitoplasmáticas) puede realizarse mediante métodos serológicos o citológicos.
Inmunocromatografía
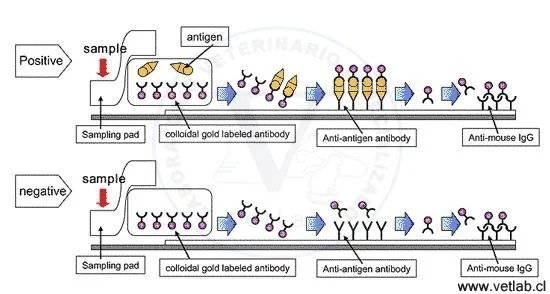

La Inmunocromatografía es un método similar a ELISA en su modalidad “sandwich”. La reacción inunológica se realiza en una membrana cromatográfica por acción capilar. Se utilizan 2 tipos de anticuerpos contra el antígeno en estudio y uno para control positivo de la reacción.
Un anticuerpo está fijado en la membrana y el otro es un anticuepo marcado con oro coloidal infiltrado en la almohadilla que absorbe la muestra. Cuando la muestra líquida es colocada sobre esta almohadilla unida a la membrana, el antígeno de la muestra forma un inmunocomplejo con el anticuerpo marcado.
Este complejo migra en la fase líquida y se pone en contacto con el anticuerpo fijado en la membrana formando otro complejo que queda fijado en la membrana, formando una línea de color púrpura que denota la presencia del antígeno en una reacción positiva.
El excedente de antiecuerpos marcados con oro coloidal continua avanzando por capilaridad para unirse a los anticuerpos anti IgG fijados en el otro extremo de la membrana coloreándose como indicador de control de la reacción.
Microscopía de cuerpos de inclusión CDV en células sanguíneas y conjuntivales
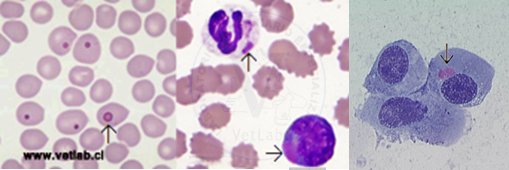
Las inclusiones suelen ser fácilmente observadas en frotis teñidos con Diff Quick.
La observación de la capa leucocitaria puede mejorar la probabilidad de encontrar estas células con inclusiones en sangre.
Dependiendo de los signos clínicos y de la etapa de la infección, las inclusiones intracitoplasmáticas se pueden encontrar en algunas células epiteliales de la conjuntiva, vejiga urinaria y en células del FCE.
El serodiagnostico, con determinación de anticuerpos IgM e IgG anti CDV, es el método diagnóstico más solicitado al laboratorio. Si bien son pruebas confiables, es preciso una adecuada interpretación.
La medición de anticuerpos séricos IgM (contra las proteínas del núcleo viral NP y P) y los IgG (contra los antígenos de la cápsula H y F) ayudan al diagnóstico del Distemper, pero no es posible discriminar los anticuerpos pasivos maternos, los anticuerpos vacunales y los anticuerpos por infecciones subclínicas, de los que son producto de la enfermedad en cachorros, en animales previamente inmunizados o que han tenido contacto previamente con el virus.
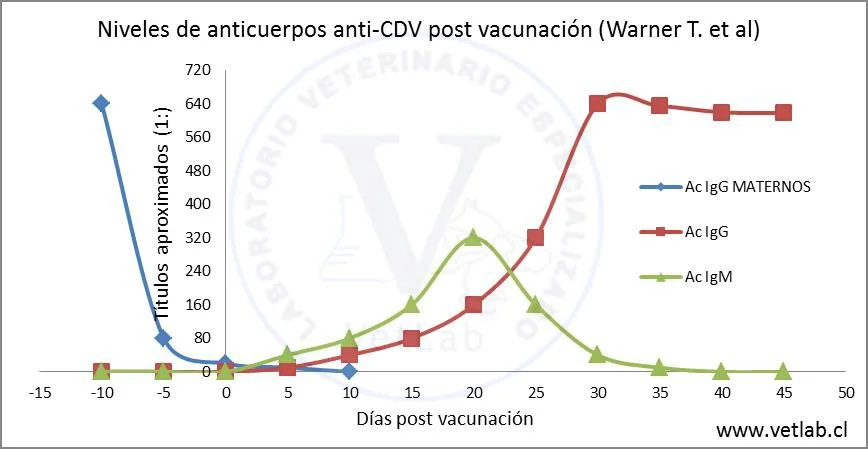
Los de tipo IgM puede ser detectados en perros infectados no vacunados, entre los 6 y 8 días post infección. Los IgG aparecen entre los 10 y 20 días.
La pruebas de inmunofluorescencia indirecta y ELISA, para la detección de IgM específica contra CDV, son pruebas útiles para el diagnóstico, ya que IgM persiste en perros infectados 1 a 3 meses, dependiendo de la cepa viral y de la respuesta del huésped.
En perros vacunados la IgM persiste por unas 3-4 semanas. Falsos negativos pueden observarse en perros que mueren en forma aguda, sin la presencia de respuesta inmunitaria, y puede darse además, en presentaciones subagudas o crónicas.
Los títulos seriados de IgG determinados en 2 muestras con 2 semanas de diferencia, son de valor en perros que no han sido vacunados dentro del mes anterior. Un aumento de cuatro veces o mas, entre la etapa de la fase aguda y la convaleciente, es señal de una enfermedad activa.
Los títulos de anticuerpos en muestras de FCE, mayores que los del suero, se atribuyen a una infección activa, puesto que esos anticuerpos se han producido localmente.
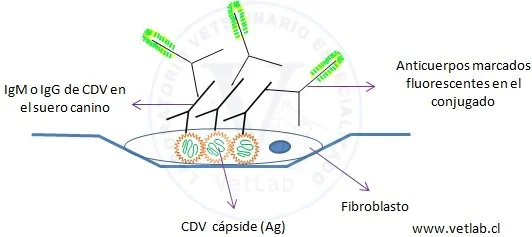
Es un método de alta sensibilidad, especificidad y valor diagnóstico, aunque su calidad es dependiente de la experiencia del microscopista. El suero o FCE canino diluido se incuba en los pocillos individuales de un portaobjeto que contiene una mezcla de células (fibroblastos) infectadas con antígenos de CDV, junto a células no infectadas.
Luego se agrega un conjugado que contiene anticuerpos anti-IgM o anti-IgG marcados con fluoresceina, los cuales se unen al complejo Ag-Ac previanente formado.
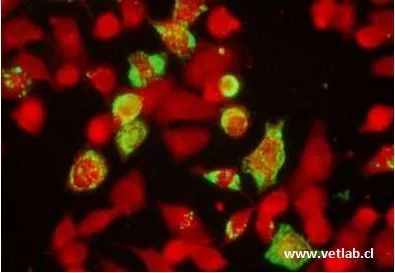
El resultado de la reacción se visualiza en un microscopio de fluorescencia en que la reacción positiva se aprecia con la emisión fluorescente color verde manzana desde la superficie de las células infectadas.
Los resultados positivos pueden ser re-testeados con diluciones mayores, para determinar el título reactivo máximo de la muestra.
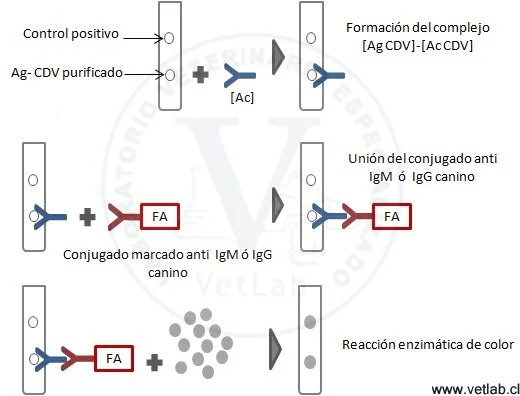
Es un método rápido, de baja complejidad y niveles intermedios de sensibilidad y especificidad para la determinación de anticuerpos anti-CDV en sangre y FCE.
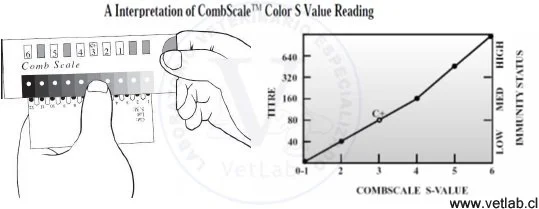
Es un ensayo inmunoenzimático indirecto. La fase sólida es un peine con 12 proyecciones (“dientes”). Cada diente está sensibilizado en dos áreas reactivas:
Punto superior — inmunoglobulina canina (Control Interno).
Punto inferior — antígeno estructural de CDV.

La Bandeja de Desarrollo tiene 6 filas (A-F) de 12 pocillos cada una. Cada fila contiene una solución reactiva lista para ser usada en cada etapa del ensayo, pasando el peine de una fila a otra, con un período de incubación en cada etapa.
La reacción coloreada positiva en la fase sólida puede cuantificarse, comparando la intensidad del color de la reacción en una escala cromática incluida en el kit (Comb Scale) e interpolando en el gráfico adjunto para estimar los títulos de anticuerpos anti-CDV.
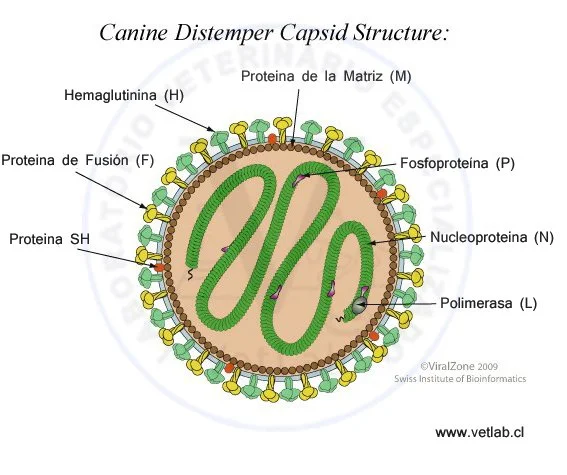
El Distemper canino es causado por un Morbillivirus perteneciente a la familia Paramyxo-viridae estructuralmente relacionado con el virus Sarampión.
El virus tiene una envoltura de RNA en una cadena de polaridad negativa y se considera un serotipo único, con cepas que varían en su tropismo y virulencia.
La secuencia de sus genes (hemaglutinina, neuraminidasa, proteína de fusión), sugieren que existen diferencias en los genotipos, condición que es de importancia para el diagnóstico molecular y para la elaboración de vacunas.
El virus se une a los receptores de la superficie celular de la célula huésped a través de la glicoproteína H (hemaglutinina).
Se fusiona con la membrana plasmática y el RNA de la cápside, para liberarse posteriormente en el citoplasma.
La replicación se inicia cuando la nucleoproteína está en cantidad suficiente para la neo-sintetisis de antigenomas y genomas virales.
El RNA de la cápside interactúa con la proteína de la matriz debajo de la membrana plasmática y de las yemas para la posterior liberación del virión.
Los métodos antes revisados para el diagnóstico ante-mortem de distemper son altamente dependientes del estadío de la infección, de la condición clínica del paciente y no siempre cumplen con los requerimientos de sensibilidad y especificidad.
Los recientes desarrollos de las técnicas de biología molecular revelan la utilidad de estos métodos con propósitos diagnósticos, como también para estudios patológicos y epidemiológicos.
La Reación de la Polimerasa en Cadena por transcripción reversa (RT-PCR) para la detección del RNA viral del CDV representa un método diagnóstico antemortem, altamente sensible y específico, temprano y seguro, utilizando muestras de sangre, suero y fluído cerebroespinal, independiente de los hallazgos clínicos, histopatológicos, de los títulos de anticuerpos y de la distribución del antígeno viral, en casos agudos y crónicos.
La RT-PCR como prueba diagnóstica es capaz de amplificar fragmentos del ácidos nucleicos en forma exponencial, aún encontrándose en la muestra una sola molécula, indetectable por otros métodos, logrando determinar la infección desde el segundo día de iniciada.
Generalmente se amplifican fragmentos del gen que codifica la nucleoproteina (NP), una región conservada dentro del genoma viral. Además, puede aplicarse al estudio de variaciones genéticas del virus, mediante caracterización molecular de secuencias claves del genoma.
Este virus también ha sido detectado en la sangre de los cachorros clínicamente normales hasta 10 días después de la vacunación con vacunas de virus atenuados, por lo que se debe tener cuidado en la interpretación de los resultados de estos pacientes.
La sensibilidad del método depende de la muestra utilizada, de la técnica de recolección, del estadio de la enfermedad, del método de extracción de ácidos nucleicos, de los cebadores (partidores o primers) y del método de PCR. Se mejora aún más la sensibilidad analítica si se utiliza la capa leucocitaria para la extracción de RNA.
Un primer (partidor, cebador, iniciador), es una cadena de ácido nucleico que sirve como punto de partida para la replicación del DNA.
Es una secuencia corta que contiene un grupo 3’hidroxilo libre, que forma pares de bases complementarios a una hebra molde y actúa como punto de inicio para la adición de nucleótidos con el fin de copiar la hebra molde.
La secuencia y posición de los nucleótidos que conforman el primer se relaciona directamente con la especificidad del fragmento de DNA que se intenta replicar.
Se necesitan dos primers para la reacción de PCR, uno en el extremo 3′ y el otro complementario para la otra hebra y se componen de aproximadamente 20 nucleótidos.
Fuentes de información para el diseño de primers:
- Electronic PCR (Natl Center for Biotechnology Information). Prueba in silico primers a utilizar en genoma humano y otros Eucariontes.
BLAST algorithm (Natl Center for Biotechnology Information). Determina si la secuencia del primer a utilizar es correcta. Útil para cualquier organismo procarionte y eucarionte.
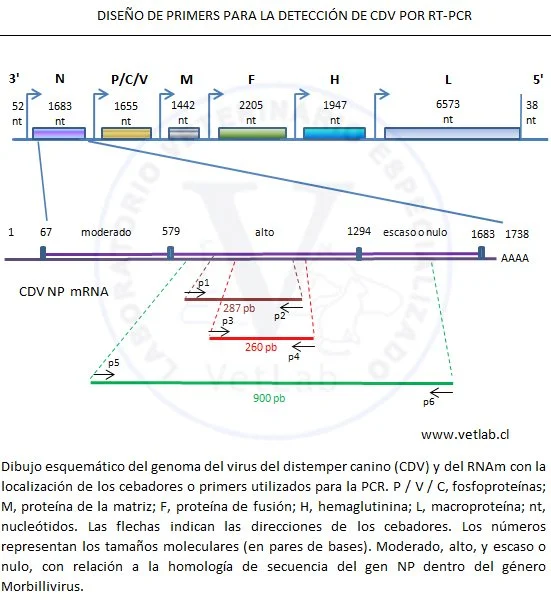
FUENTE: J Clin Microbiol. 1999 November; 37(11): 3634–3643.
La muestra de sangre debe ser conservada en EDTA hasta por 5 días. Mediante centrifugación de esta sangre, se sapara la capa de leucocitos (buffy coat) que es el material utilizado para la extracción del RNA del virus distemper. El volumen de sangre es importante, para aumentar la cantidad de RNA aislado obtenido de la muestra.
El método de extracción se basa en la lisis de las células y de la cápside viral para la liberación del mRNA contenido en ésta y la separación del del RNA con solventes. El RNA presente en la fase acuosa se purifica por adsorción en columna de matriz para RNA. La concentración de RNA obtenido en la solución final puede determinarse por espectrofotometría a 296nm.
MUESTRA: SANGRE CON EDTA > 1 mL
EXTRACCIÓN DEL RNA
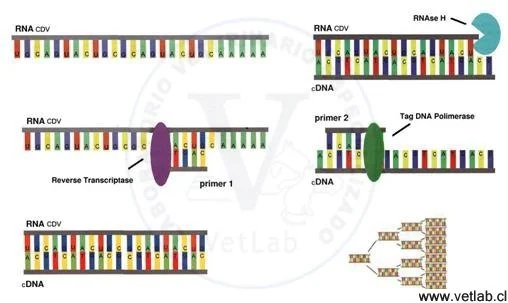
El mRNA purificado de la muestra es utilizado posteriormente para la síntesis de la primera cadena de cDNA mediante Transcripción Reversa. En este proceso intevienen el RNA aislado (molde), la enzima Transcriptasa Reversa (de leucemia murina), cebadores de DNA, RNAasa, desoxinucleótidos trifosfato (dNTP) y buffers.
El primer 1 se une a la cadena de RNA, y mediante la actividad de la Transcriptasa Reversa se extiende una cadena cDNA, resultando una molécula híbrida. A continuación, mediante la actividad de la enzima RNAasa H, se degrada el RNA viral que fué utilizado como molde.
El primer 2 se une al cDNA monocatenario para sintetizar la hebra complementaria y se procede a la correspondiente elongación mediante la actividad de la DNA polimerasa.
La amplificación del DNA que se ha sintetizado, se consigue mezclándolo con los primers de complentaridad específica al extremo 3´ del DNA, los cuales actuan como iniciadores para la actividad de la polimerasa. En esta solución deben también estar presentes los dNTPs que son las unidades que componene el DNA, en una mezcla equimolar, y la TAq polimerada (enzima termoestable aislada de la bacteria Thermus aquaticus).
Esta síntesis exponencial de DNA por RT-PCR incluye 3 pasos:
1. Desnaturalización o separación de las hebras del DNA (94°C x 1,0 min)
2. Alineamiento o apareamiento de los primers a la región complementaria del DNA (59,5°C x 2,0 min)
3. Extensión o síntesis del nuevo DNA idéntico, mediante la actividad de la polimerasa (72°C x 1,0 min)
Estos pasos se repiten automáticamente unas 40 veces en un termociclador, para crear millones de copias del fragmento del DNA, derivado de la transcipción del RNA del virus distemper.
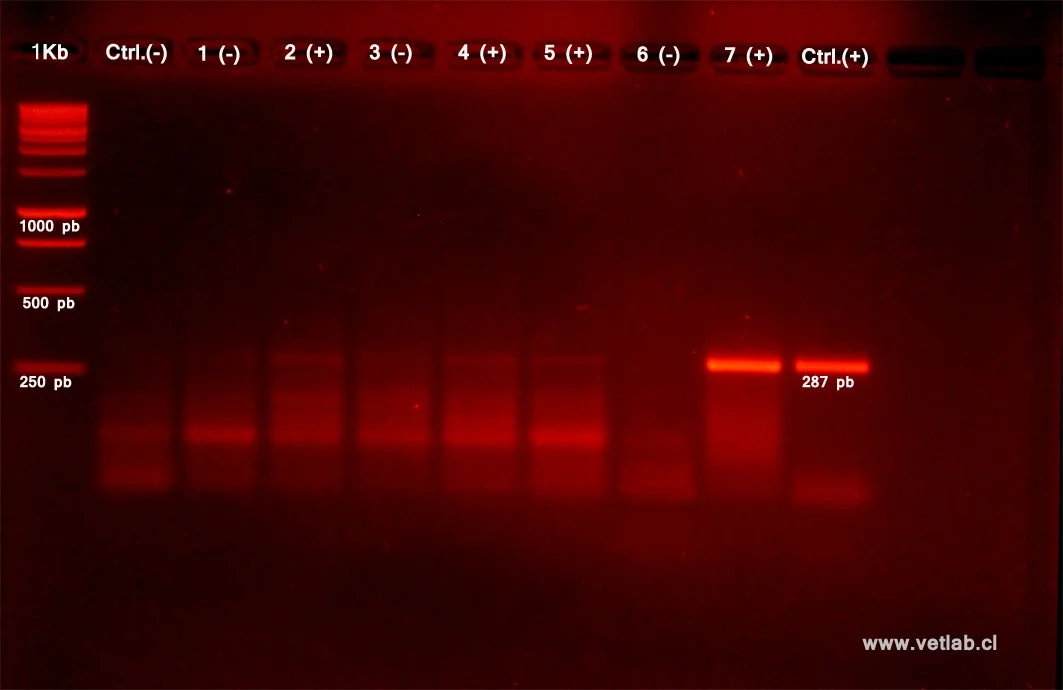
El resultado del DNA amplificado se analiza mediante electroforesis en gel de agarosa al 1,5%, previamente cargado con muestras y controles, bajo un campo eléctrico de 60v durante 15 min. El gel luego es teñido con Bromuro de Etidio y se observa y fotografía en un visualizador de electroforetograma.
Los canales electroforéticos son cargados con un control negativo (muestra libre de DNA), un control positivo (muestra con antígeno viral confirmado), marcadores de peso molecular y los productos PCR de cada muestra analizada. El resultado de esta PCR convencional es expresado en forma cualitativa (positivo o negativo).
Está previamente definido que el DNA transcrito del RNA del CCV migra a una distancia equivalente a 287 pb. Confirmación que se hace por comparación con el marcador de peso molecular desplegado en el elecroforetograma.